Family SSU_rRNA_bacteria
Download the aligments for this family. (.stk)
RNA Shape
Level1
()((((((((((())))(((((())()())))(((())))(()))()())))((()))))(())(((())))((()))))((((((((())))(((((()))(())))))))((()))(()())))(())((((((((()(((()()))(((((((()())(((()))(()))))))))))))(((())))(()))()))))((((((((()))))))))()
Level2
()(((()((()()())()())()())())()()())((()(()()))()(()()))()(((()((()())((()())(()()))))()())())()()
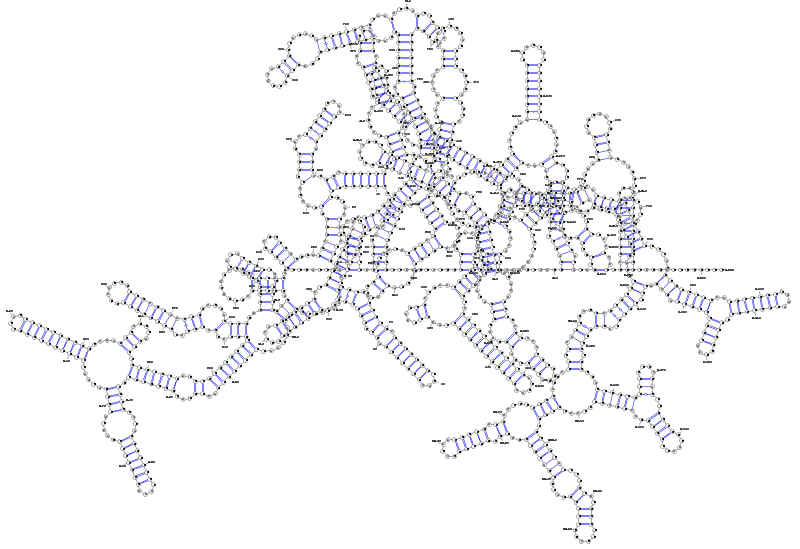
Consensus Secondary Structure representation
>predicted reference SSU_rRNA_bacteria
UUUAUGGAGAGUUUGAUCCUGGCUCAGGGCGAACGCUGGCGGCGCGCUUAACACAUGCAAGUCGAGCCCCACGGGGCCUUAAGGCCCCGAGGGGGCGGCGAACGGGUGAGUAACGCGCGGGCAACCUACCCCCGGGUGCGGGAUACCCCCCGGAAACGGGGGGUAAUACCGCAUAAUGCCCCGGCCCGUAUGGGCCGGGGCUAAAGGCAUUUAGCCGCCCGGGGAUGGGCCCGCGCCCCAUCAGCUAGUUGGUGGGGUAAUGGCCCACCAAGGCGAUGAGGGGUAGCCGGCCUGAGAGGGCGAUCGGCCACACUGGGACUGAGACACGGCCCAGACUCCUACGGGAGGCAGCAGUGGGGAAUAUUGCGCAAUGGGCGAAAGCCUGACGCAGCGACGCCGCGUGGGGGAUGAAGGCCUUCGGGCCGUAAACCCCUUUCCCGGGGGAAGAAAAACCGAUAUUUUAUCGGUUUGACGGUACCCCGGGAAGAAGCCCCGGCUAACUCCGUGCCAGCAGCCGCGGUAAUACGGAGGGGGCAAGCGUUGCCCGGAAUUAUUGGGCGUAAAGGGCGCGCAGGCGGCCCGGCAAGUCGGGCGCGAAAUCCCGCGGCUCAACCGCGGGAUCGCGCCCGAAACUGCCGGGCUUGAGUCCGGCCGAGGGGGGCGGAACUCCCGGUGUAGCGGUGAAAUGCGUAGAUAUCGGGAGGAACACCACUGGCGAAGGCGGCCCCCUGGGCCGGUACUGACGCUGAGGCGCGAAAGCGUGGGGAGCAAACGGGAUUAGAUACCCCGGUAGUCCACGCCGUAAACGAUGGGCGCUAGGCGCCGGGGGGAUUAACCCCCCGGCGCCGAAGCUAACGCAUUAAGCGCCCCGCCUGGGGAGUACGGCCGCAAGGCUGAAACUCAAAGGAAUUGACGGGGGCCCGCACAAGCGGUGGAGCAUGUGGUUUAAUUCGAUGCAACGCGAAGAACCUUACCCGGGCUUGACGUGCCGGGAACCCCCCGGAAACGGGGGGUCCCCUUAAGGGGCCCGGACACAGGUGCUGCAUGGCUGUCGUCAGCUCGUGUCGUGAGAUGUCGGGUUAAGUCCCGCAACGAGCGCAACCCCCGCCCCUAGUUGCCAUCGGUUUAUUCCGGGCACUCUAGGGGGACCGCCGGCGAUAAGCCGGAGGAAGGUGGGGAUGACGUCAAGUCAUCAUGGCCCUUAUGCCCGGGGCUACACACGUGCUACAAUGGCCGGCACAAAGGGUAGCAAAGCCGCGAGGCGGAGCUAAUCCCAUAAAGCCGGCCUCAGUUCGGACUGGAGGCUGCAACUCGCCUCCAGGAAGUUGGAAUCGCUAGUAAUCGCGGCUCAGCAUGCCGCGGUGAAUACGUUCCCGGGCCUUGUACACACCGCCCGUCACGCCACGGAAGCCGGCCCCGCCCGAAGCCGCCGCGCCAACCUUAAGGAGGCGGGCGGCUAAGGCGGGGCCGGCGACCGGGGCGAAGUCGUAACAAGGUAGCCGUACCGGAAGGUGCGGCUGGAUCACCUCCUUU
........(((((.......))))).((((.((((((.(((((((((....(((.(((..(((..(((((.(((((((....))))))).)).))))))......(((......((((((((..((...(((((((.((((....(((((((....))))))).....)))).....((((((((((....))))))))))....(((.....))).)))))))..))))))))))(((..(.(((..((((((((.......))))))))))).....))))..((((((((....))))...))))))).(((((............))))).((((....))))...)))))).).....(.(((...(((((....))))).)))).)).))))))..((((......((((....)))).....))))...(((((((...(.....(((.........))).....)....))))))...)..(((((......(((((.....((....)).......)))))))))).))))))))))..........(((.....(.((((.(.(((.(((((((.(((((((((((....(((((((.....)))))))..)))))))))..)))))))))...(((((((((..((((((((...((((((((...(((......)))......))))))))...)....(..((....)))))))))).)))))).)))...))))..))))....((((((...((...((((.........))))...))))))))..........((((((..((((((((((((.....))))))))))))...((....)).....)))))))))).(((......((((....))))....)))........(((((.(((((((.((..((((((.((((((((((....((((........))))........(((((((.....((((((((..(((((((....))))))).((((....))))))))).))).((.((((..((((((.((...(((((((((....)))..((((......))))..)))))).....((((.(((((((...((..(((......)))))....)))))))..((.(((((.....))))).)).....))))....)).).)))...))))))))....)))))))...)).)))))))).)...(((((((.....(((..((...(((....)))...))....))).....)))))))......(...((((((((........))))))))...).....))))).....((((((((.......))))))))......))...)))))))))).))..(.(..((...((((.(((..((((((((((((....((((((.((((..((....)).))))))))))...))))))))))))..))).))))....))...)..).((((((((((....)))))))))).............